Genom- und Populationsdynamik während der chronischen Infektion mit Helicobacter pylori
Lorem ipsum dolor sit amet, consetetur sadipscing elitr
Die Infektion mit dem Magenbakterium Helicobacter pylori gehört zu den häufigsten Infektionen überhaupt, mehr als die Hälfte der Weltbevölkerung sind mit diesem Bakterium infiziert. Die Infektion wird oft im Kindesalter erworben und besteht dann jahrzehntelang fort, so dass die H. pylori-Infektion ein besonders charakteristisches Beispiel einer chronischen Infektion anzusehen ist, die ja das Rahmenthema des SFB sind. Längst nicht alle mit H. pylori infizierten Personen erleiden aufgrund der Infektion Folgeschäden: bei ca. 85 Prozent der Infizierten bleiben Symptome aus. Etwa 10-15 Prozent bekommen ein Magen- oder Zwölffingerdarmgeschwür und etwa 1 Prozent der Infizierten entwickeln die gefährlichste Komplikation der H. pylori-Infektion, ein Magenkarzinom. Aufgrund der hohen Zahl der Infizierten, ist H. pylori damit für ca. 800.000 neue Krebsfälle pro Jahr ursächlich verantwortlich.
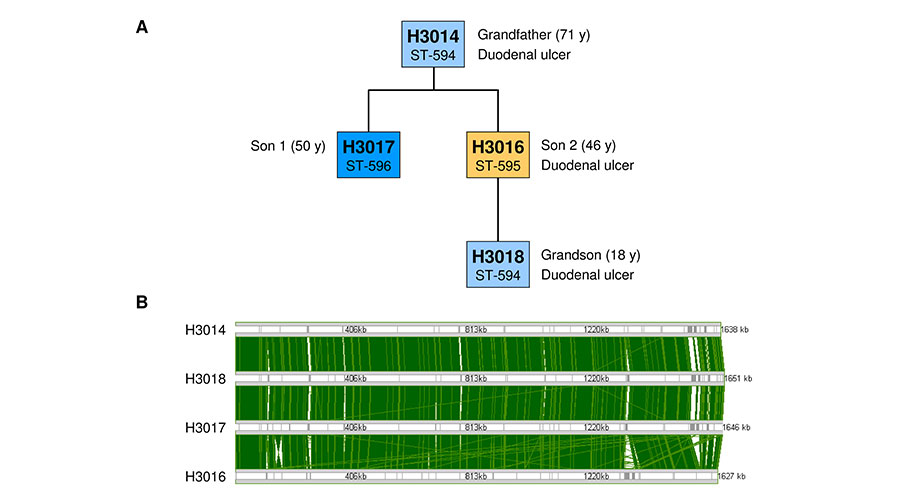
Schon seit etwa 30 Jahren weiß man, dass sich die H. pylori-Bakterien unterschiedlicher Patienten voneinander unterscheiden. Jeder Patient hat seinen eigenen oder sogar mehrere individuelle H. pylori-Bakterien, so wie jeder Mensch seinen eigenen charakteristischen Fingerabdruck hat.
Die Forschergruppe um Sebastian Suerbaum arbeitet an der Aufklärung der molekularen Mechanismen, die die extrem hohe genetische Vielfalt von H. pylori schaffen, und will die Frage klären, wie die hohe Variationsfähigkeit dieser Bakterien zum Infektionsverlauf beiträgt.
Wissenschaftliche Vorgehensweise
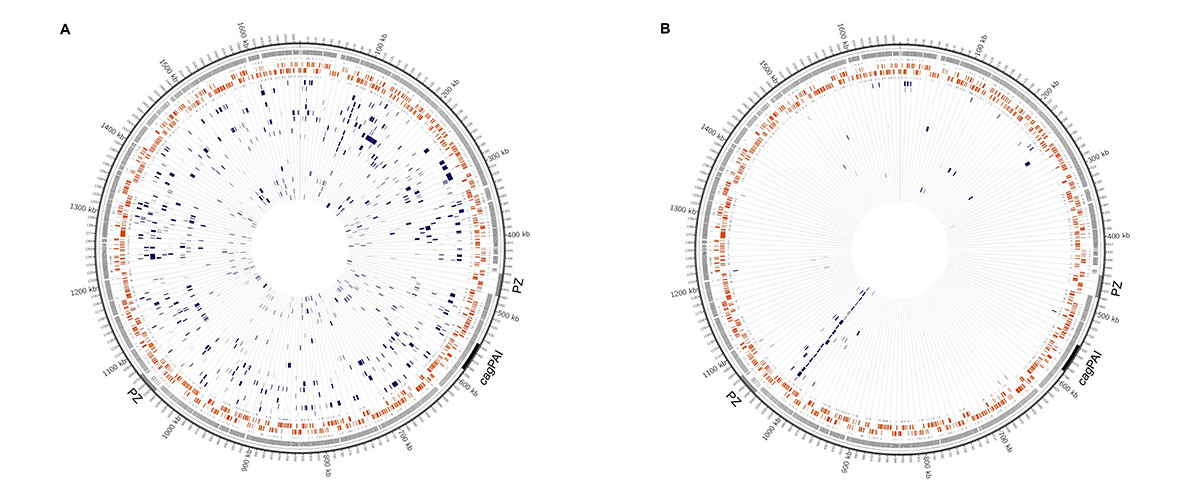
Die Forschergruppe verwendet verschiedene Hochdurchsatz-Sequenzierungstechnologien („Next Generation Sequencing“), um die Genomsequenzen von H. pylori-Bakterien zu entschlüsseln. Um die Mechanismen der genetischen Diversität zu verstehen, werden einerseits Isolate von verschiedenen Patienten und von verschiedenen Bereichen des Magens analysiert.
Weiterhin vergleichen wir Isolate, die im Verlauf mehrerer Jahre von den selben Patienten isoliert wurden, um zu prüfen, inwieweit sich die Bakterien an den individuellen Träger anpassen. Für die Analyse vieler verschiedener Bakterienstämme wird die Illumina-Technologie (MiSeq) verwendet, bei der die Genome in durchschnittlich 25 Stücken (Contigs) vorliegen. Die Single Molecule, Real-Time-Technologie (SMRT-Sequencing, Pacific Biosciences) erlaubt es, die Genome vollständig zu sequenzieren, so dass das komplette ringförmige Chromosom sequenziert wird, aufgrund höherer Kosten und geringeren Durchsatzes können aktuell aber nur ausgewählte Stämme mit dieser Technologie sequenziert werden – die Kombination der beiden Methoden ermöglicht die Sequenzierung einer großen Zahl von Isolaten, bei denen ausgewählte Stämme als Referenzgenome fungieren.
Die Genomsequenzierung wird im Projekt mit experimentellen Zugängen kombiniert. Es werden Mutanten konstruiert, bei denen einzelne H. pylori-Gene inaktiviert wurden und die Auswirkungen dieser Mutationen auf die Fähigkeit der genetischen Diversifizierung gemessen. So sollen die Mechanismen der genetischen Diversifizierung auf molekularer Ebene entschlüsselt werden. Ein weiteres Thema des Projekts ist die bakterielle Epigenetik. Die SMRT-Sequenzierung erlaubt es, im Genom der Bakterien Basen zu identifizieren, die durch Methylierung modifiziert sind. H. pylori-Stämme verändern im Verlauf der chronischen Infektion ihre Methylierungsmuster und die Arbeitsgruppe erforscht die Mechanismen dieser Methylierung und die Auswirkungen von Veränderungen des Methylierungsmusters.
Lorem ipsum dolor sit amet, consetetur sadipscing elitr
Sebastian Suerbaum erzählt über seine Foschung im SFB 900
Sebastian Suerbaum ist seit Gründung des SFBs im Jahr 2010 mit dabei. Sein Teilprojekt A1 widmet sich der Erforschung der genetischen Variabilität von H. pylori und dem Zusammenhang dieser genetischen Vielfalt und dem chronischen Infektionsverlauf. Des Weiteren betreut Prof. Suerbaum zusammen mit Prof. Tümmler das Projekt Z1, in welchem Hochdurchsatz-sequenzierungen und Bioinformatik als Serviceleistungen für den ganzen SFB angeboten werden. Prof. Suerbaum ist außerdem stellvertretender Sprecher im SFB.
Publikationen des Forschungsprojektes A1
Kontakt

Prof. Dr. med. Sebastian Suerbaum
Vorstand Medizinische Mikrobiologie und Krankenhaushygiene
Max von Pettenkofer-Institut
Ludwig-Maximilians-Universität München
Pettenkoferstr. 9a
80336 München
+49 89 2180-72800
suerbaum@mvp.uni-muenchen.de