Chronische Pseudomonas aeruginosa Infektionen in Patienten mit Mukoviszidose: Mikroevolution des Erregers und genetische Prädisposition des Wirts
Pseudomonas aeruginosa auf Blutagar-Platte
Patienten mit Mukoviszidose (auch Cystische Fibrose (CF)) leiden an einer Fehlfunktion der exokrinen Drüsen. Der Ausfall oder die Einschränkung der Funktion eines bestimmten Transporterproteins beeinflusst die Zusammensetzung von Sekreten, die in Magen, Darm, Bauchspeicheldrüse und auch in den Atemwegen produziert werden. Die Sekrete sind übermäßig zähflüssig und verursachen Funktionsstörungen in den betroffenen Organen. In den Atemwegen führt dies unter anderem zu einer Austrocknung der Schleimhäute, die so anfälliger für bakterielle Infektionen werden. Bei 80 % der erwachsenen CF-Patienten sind die Atemwege dauerhaft mit P. aeruginosa besiedelt. Vom Immunsystem der Patienten kann dieses Bakterium nicht vollständig entfernt werden und verursacht chronische Infektionen, durch die das Gewebe in den Atemwegen geschädigt und die Lungenfunktion drastisch vermindert. Bei insgesamt 90 % der CF-Patienten bestimmt die chronische Infektion mit P. aeruginosa so den klinischen Zustand der Patienten und den weiteren Verlauf der Krankheit.
Ziel des Projekts ist es zu analysieren, inwieweit Genomvariationen sowohl auf Seiten des Pathogens als auch auf der Wirtsseite zu den teilweise sehr unterschiedlich schweren Krankheitsverläufen beitragen. Bei P. aeruginosa spielen dabei nicht nur Charakteristika der initial besiedelnden Stämme eine Rolle, sondern auch während der chronischen Kolonisation neu auftretende Genomvariationen. Diese Mikroevolution in den CF-Atemwegen kann bakterielle Phänotypen modifizieren und trägt anscheinend zur Adaptation des bakteriellen Lebensstils an das Habitat CF-Lunge bei sowie zu vermehrter Resistenz gegen Mechanismen der Immunabwehr und auch gegen Behandlungen mit antimikrobiellen Substanzen. Daher sollen langfristig in diesem Projekt sowohl genetische Variationen der Patienten als auch die Mikroevolution der Bakterien während der chronischen Infektion analysiert werden und nach möglichen Assoziationen zwischen beiden gesucht werden. Die Identifikation von CF-spezifischen bakteriellen Mikroevolutionsprozessen und die damit verbundenen Adaptationsstrategien können Ansatzpunkte für neue Behandlungsstrategien von chronisch infizierten CF-Patienten liefern.
Wissenschaftliche Vorgehensweise
Zur Aufklärung des detaillierten Anpassungsprozesses von P. aeruginosa an die Lunge von Mukoviszidosepatienten steht an der Medizinischen Hochschule Hannover eine einzigartige Stammsammlung zur Verfügung. Diese umfasst sequentielle P. aeruginosa Isolate von verschiedenen Mukoviszidosepatienten, die seit Beginn der Besiedlung halbjährlich archiviert wurden. Somit ist ein Langzeitrückblick, in einigen Fällen über mittlerweile bis zu 30 Jahren Kolonisationszeit, möglich und es besteht erstmals die Möglichkeit, den gesamten bakteriellen Adaptationsprozess an Patienten mit unterschiedlichem Krankheitsverlauf zu untersuchen und anschließend zu vergleichen.
Von insgesamt zwölf Mukoviszidosepatienten, sechs mit mildem und sechs mit sehr schwerem Krankheitsverlauf, wurden sequentielle Isolate in Einjahresintervallen ausgewählt, so lange der erste initiale Klon nachgewiesen werden konnte. Diese Isolate wurden sowohl Genom-sequenziert als auch ausgiebig phänotypisch charakterisiert.
Aus NGS-Datensätzen wurden die genomischen Variationen (SNPs, Indels, Leserasterverschiebungen, Stopmutationen, große Deletionen, Aufnahme fremden genetischen Materials) ermittelt, die im Laufe der chronischen Infektionen aufgetreten waren. Durch Vergleich der zwölf Verläufe wurden die genomischen Bereiche bestimmt, die eine erhöhte Mutationsfrequenz aufwiesen (sogenannte „Hot-spots“) und somit potentiell mit der Diversifizierung und/oder einer positiven Selektion im Habitat CF-Lunge assoziiert sind.
Außerdem wurden anhand der genomischen Variationen für jede der zwölf Isolat-Reihen phylogenetische Bäume erstellt und damit der jeweilige Verlauf der Mikroevolution beschrieben. Insgesamt sollen die Ergebnisse auf sowohl auf das Vorkommen CF-spezifischer Merkmale der Mikroevolution als auch auf Unterschiede zwischen milden und schweren Krankheitsverläufen hin untersucht werden.
Kompetitive Fitnessexperimente sollen zudem Aufschluss über die relative Fitness der einzelnen Isolate in nährstoffreichen als auch nährstoffarmen unbelebten Habitaten geben und die unterschiedlich ausgeprägten Folgen der Adaptationsprozesse in einem Wirtshabitat mit einem hoch aktivierten Immunsystem für die Fitness zulassen.
Diese Wachstumsexperimente können außerdem Rückschlüsse darauf zulassen, inwieweit Adaptation und Spezialisierung die Fähigkeit der Bakterien beeinträchtigen, auch neue, veränderte Habitate zu besiedeln und dort zu bestehen. Für diese Fitnessexperimente werden die jeweils sequenzierten sequentiellen Isolate eines Langzeitverlaufs unter nährstoffarmen bzw. nährstoffreichen Bedingungen zusammen über einen längeren Zeitraum kultiviert. In regelmäßigen Abständen werden Proben entnommen, in denen die relativen Anteile der einzelnen Isolate aus Amplikon-Sequenzierungsdaten bestimmt werden, indem Isolat-spezifische genomische Variationen, die durch die vorherigen Genomsequenzierungen detektiert wurden, als Marker verwendet werden.
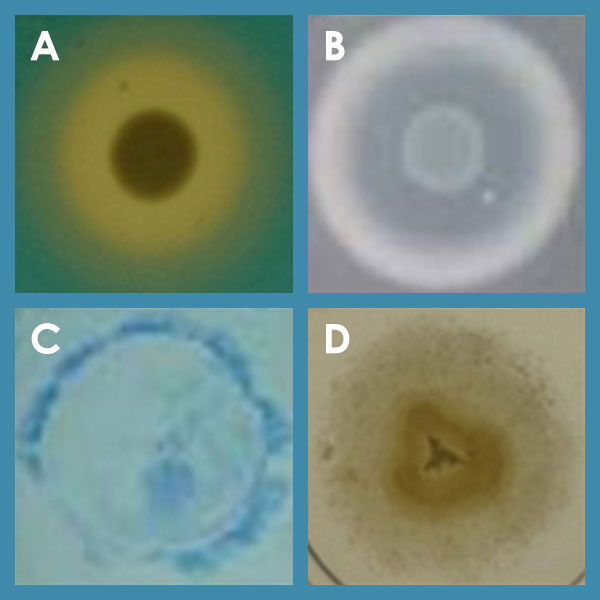
Einige Phänotypen von P. aeruginosa; A: Sekretion von Siderophoren; B: Sekretion von Proteasen; C: Twitching motility; D: Swimming motility
Des Weiteren dazu wurden in dem Forschungsvorhaben genomweite Sequenzanalysen von 100 Isolaten global dominierenden P. aeruginosa Klone C40A und PA14 sowie Amplikonsequenzierungen für Isolate aus anderen Mukoviszidose- Zentren und aus anderen Habitaten durchgeführt. Die Genomsequenzierungen der 100 Isolate diente zur Bestimmung der intraklonalen Diversität. Diese Isolate wurden auf individuelle oder weiter verbreitete Genomvariationen hin untersucht und für ausgewählte Isolate wurde ebenfalls die kompetitive Fitness bestimmt.
Der zweite Ansatz diente zu Untersuchung des globalen Auftretens der Mikroevolutionsereignisse, die für die sequentiellen CF-isolate gefunden worden waren. Die dort bestimmten Mutations-Hotspots sowie weitere Gene, die laut Literatur an der Adaptation von P. aeruginosa an die CF-Lunge beteiligt sind, wurden per Amplikonsequenzierung in Isolaten aus anderen Stammsammlungen untersucht. Diese Sammlungen umfassten sowohl CF-Isolate aus anderen Kliniken als auch aus anderen Habitaten (Isolate von COPD-Patienten, von akuten Infektionen, Umweltisolate). Auf diese Weise wurde das globale Mutationsspektrum in diesen >30 Genen bestimmt, und die Mutationen konnten als individuell oder Habitat-spezifisch charakterisiert werden.
Isolate mit potentiell vermeintlich funktionell bedeutsamen Mutationen sollen anschließend auf ihre Fitnesscharakteristika, auf veränderte Funktionen der in den betroffenen Gene kodierten Proteine und auf dadurch bedingte phänotypische Änderungen hin getestet werden.
Der Einfluss des genetischen Hintergrundes der Mukoviszidose-Patienten aud den Verlauf der chronischen P. aeruginosa-Infektion soll durch Genomsequenzierungen der 12 Patienten analysiert werden, aus deren Atemwege die im Detail analysierten sequentiellen Bakterienisolate stammten. Potentielle genomische Variationen, die mit milden oder schweren Krankheitsverläufen assoziiert sind, werden als Kandidaten für Modulatoren des Schweregrads des klinischen Verlaufs angesehen, deren Auftreten bei anderen CF-Patienten bestimmt werden sollen. Auf diese möglichen Modulatoren hin sollen große Kohorten von Patienen getestet werden, von denen DNA-Proben in der „European-Canadian unified CF DNA Biobank“ hinterlegt sind, Proben von mehreren Tausend CF-Patienten aus Deutschland, der Schweiz und Kanada. Potentiellen Modulatoren, deren Rollen in dieser großen Patienten-Kohorte bestätigt werden können, könnten dann als neue Biomarker für den Schweregrad der Verlaufs einer chronischen P. aeruginosa-Infektion in CF-Patienten darstellen.
Burkhard Tümmler erzählt über seine Forschung im SFB 900
Burkhard Tümmler ist seit Gründung des SFBs im Jahr 2010 mit dabei. In seinem Teilprojekt A2 wird die Evolution von Pseudomonas aeruginosa während einer chronischen Infektion in Patienten mit Mukoviszidose untersucht. Des Weiteren betreut Prof. Tümmler zusammen mit Prof. Suerbaum das Projekt Z1, in welchem Hochdurchsatz-sequenzierungen und Bioinformatik als Serviceleistungen für den ganzen SFB angeboten werden.
Publikationen des Forschungsprojektes A2
Kontakt

Prof. Dr. med. Burkhard Tümmler
Klinische Forschergruppe – OE6711
Klink für Pädiatrische Pneumologie, Allergologie und Neonatologie
Medizinische Hochschule Hannover
Carl-Neuberg-Str. 1
30625 Hannover
+49 511 532-2920
+49 511 532-6723
Tuemmler.Burkhard@mh-hannover.de